INTRODUCCIÓN
Tetrahymena es un género monofilético de protozoos ciliados con 41 especies descritas, muchas de las cuales son morfológicamente indistinguibles, siendo generalmente consideradas como morfoespecie (Lynn y Doerder, 2012; Xiong et al., 2019). Algunas especies de este género se utilizan como modelos biológicos en el análisis genómico, ya que son fáciles de cultivar y por el hecho de que cualquier gen puede modificarse mediante reemplazo dirigido (Wilkes et al., 2009; Ruehle et al., 2016). Entre estas especies se encuentra Tetrahymena hegewishi, que se utiliza ampliamente en proyectos asociados con la composición y evolución del ADNr y la caracterización de metalotioneínas (Nielsen et al., 1985; Schlegel, 1991; Van de Peer et al., 1993; Coyne y Yao, 1996; Ye y Romero, 2002; Jung et al., 2011; Chakraborty et al., 2014; Chang et al., 2014; Méndez-Vilas, 2014). A pesar de la importancia de esta especie de protozoos ciliados en la investigación microbiana, el comportamiento de la misma es poco conocido (Lynn y Doerder, 2012).
El cangrejo terrestre azul, Cardisoma guanhumi (Brachyura: Gecarcinidae) es una especie altamente adaptada a la vida terrestre que se encuentra comúnmente en costas, áreas de estuarios y manglares (De Oliveira-Neto et al., 2008). Se sabe que tolera grandes variaciones de salinidad, desarrollando el estadio larvario (zoea) en el agua y los otros estadios en toda la zona costera (Giménez-Hurtado et al., 2015). Su distribución va desde Bermuda hasta Florida, a través del Golfo de México, y desde las Antillas hasta el sur de Brasil (Barrios Saucedo, 2008).
Algunos autores han informado sobre eventos de depredación entre protozoos ciliados y larvas de artrópodos (Washburn et al., 1988). Muchos de estos depredadores son miembros del género Tetrahymena (Hymenostomatida: Tetrahymenidae) que, aunque son bacteriófagos, pueden presentar histofagia (principalmente facultativa), siendo algunos de ellos considerados parásitos en crustáceos (Kaczanowski et al., 2016). C. guanhumi tiene una importancia comercial relativa en República Dominicana, Estados Unidos, Brasil, Colombia y Cuba, donde se explota a gran escala y de forma artesanal (Giménez-Hurtado et al., 2015). Con relación a lo expuesto anteriormente se sabe que el impacto del ciliado en los cultivos de C. guanhumi conduce a pérdidas económicas entre los cangrejeros. Según Hwang et al. (2019) la cantidad de células en cultivos de T. hegewishi aumenta a concentraciones bajas de salinidad, por lo que el uso de cambios bruscos de salinidad plantea una alternativa contra su proliferación. El presente trabajo reporta el efecto del uso de cambios bruscos de salinidad en poblaciones de T. hegewishi en eventos de depredación contra larvas de C. guanhumi.
OBJETIVOS
- Documentar los eventos de depredación de larvas de Cardisoma guanhumi por ciliados del género Tetrahymena ocurridos en cultivos de cangrejos.
MATERIALES Y MÉTODOS
Diseño experimental. Se recolectaron zoeas de etapa uno de C. guanhumi en el Acuario Nacional de Santo Domingo. Estas se encontraban en un estanque de agua dulce con una temperatura de 28 ºC y una salinidad de 3.40 ppm. En el laboratorio, las zoeas se dividieron en 20 recipientes con 100 ml de agua dulce con diferentes valores de salinidad. El tratamiento A contó con una salinidad de 0.2 ppm, debido a que esta salinidad es el estándar en los cultivos del Acuario Nacional. El tratamiento control se basó en la salinidad del estanque de recolección de larvas, siendo esta 3.4 ppt. Los valores de salinidad se midieron con un medidor mutilparámetros APERA PC60.
Los recipientes se colocaron siguiendo un diseño de bloques completamente al azar constituyendo diez repeticiones por tratamiento. Cada recipiente contaba con tres zoeas representadas como subréplicas, ya que para el análisis estadístico se tomaron como datos los valores medios de cada recipiente . Después de 24 horas, se procedió a contar los trofontes de T. hegewishi aparecidos en un rango de 1 mm2 alrededor de todo el cuerpo de la zoea utilizando una lupa estereoscópica y un microscopio Amscope T690C.
PCR, clonación e identificación molecular de ciliados. Los análisis de PCR, clonación e identificación molecular de ciliados fueron realizados en el laboratorio de Parasitología del Instituto de Investigación en Análisis Químicos y Biológicos (IAQBUS) perteneciente a la Universidad de Santiago de Compostela, España. Para la realización de estos análisis, se extrajo y purificó el ADN de los individuos de T. hegewishi con un kit DNAeasy Blood and Tissue (QIAGEN, 2021a) de acuerdo con las instrucciones del fabricante. Se analizó el ADN para estimar su calidad, pureza y concentración, mediante la medición de A260 en un espectrofotómetro NanoDrop ND-1000 (Thermo Fisher Scientific Inc., 2010). El ADN se almacenó a -20º C hasta su uso.
Para la amplificación por PCR, se seleccionó una región de 1132 pb del gen que codifica el ARN 18 S ribosómico de subunidad pequeña (SSUrRNA) utilizando un par de cebadores universales diseñados y optimizados mediante el programa Primer 3Plus (Untergasser et al., 2007) con parámetros predeterminados. Las mezclas de PCR (25 µL) contenían buffer de reacción de PCR (Tris-HCl 10 mM, KCl 50 mM, MgCl2 1.5 mM, pH 9.0), 0.2 mM de cada desoxinucleósido trifosfato (Roche Sequencing, 2021), 0.4 µM de cada cebador [adelante 5’-TGCTTGATCCTGCCAGTAGTC-3 ‘/ inverso 5’-GATCCTTCCGCAGGTTCA-3’] (F1118S / R1118S); 0.5 unidades de polimerasa Taq de alta fidelidad (Nzytech, 2021) y 50 ng de ADN genómico. Las reacciones se realizaron en un termociclador automático (BIORAD, 2021) de la siguiente manera: desnaturalización inicial a 94º C durante 5 min, seguida de 35 ciclos a 94º C durante 30 s, recocido a 55 º C durante 45 sy 72 º C durante 1 min; y finalmente una fase de extensión de 7 min a 72º C. Los productos de la PCR se analizaron en un gel de agarosa al 1 % en tampón de ácido etilendiaminotetraacético acetato de tris (TAE) (acetato de tris 40 mM, pH 8.0, EDTA 2 mM) que contenía Sybr Green a una concentración 1X (Intron, Corea), para verificar la presencia de bandas. Los productos de PCR se clonaron en el kit de vector de clonación de ADN pSpark® II (CANVAX, 2021) de acuerdo con las instrucciones del fabricante. Después de la ligación del fragmento de PCR, las células de E. coli DH5α se transformaron y luego se seleccionaron basándose en la sensibilidad a los antibióticos y el color mediante cultivo en placas de agar LB que contenían 100 µg / ml de ampicilina, con 50 µl de una solución madre de 20 mg / ml de 5-bromo-4-cloro-3-indolil-β-galactósido (X-Gal) y 20 µl de una solución 0.5 M de isopropiltio-β-D-galactósido (IPTG) esparcidos sobre la superficie. Se amplificaron diez colonias blancas de E. coli por muestra de ligación en medio LB y se purificó el ADN plasmídico con el kit QiAaprep® Spin Miniprep (QIAGEN, 2021b) de acuerdo con las instrucciones del fabricante. A continuación, los productos amplificados por PCR se secuenciaron mediante el método de Sanger en direcciones complementarias (Sigma-Aldrich, 2021). Se utilizó la interfaz BLAST y el programa BlastN optimizado para secuencias muy similares (megablast) (U.S. National Library of Medice, 2021), para calcular el grado de identidad entre las secuencias de nucleótidos y proceder a su identificación molecular.
Análisis estadístico. Se utilizó una prueba t de Student para determinar si existe un efecto de salinidad significativo en el evento de depredación de trofontes de T. hegewishi a larvas de C. guanhumi. La temperatura no fue considerada dentro del análisis estadístico ya que cumple con el rango de tolerancia mencionado por Nyberg (1981). Todos los análisis se realizaron con un nivel de significancia de 0.05, utilizando el programa SPSS Statistics (IBM 2017).
RESULTADOS
Según el análisis de identificación molecular, se confirmó que el ciliado depredador es T. hegewishi. Después de 36 horas del avistamiento del primer grupo de trofontes de T. hegewishi, todas las zoeas estaban muertas en ambos tratamientos (Fig. 1). Algunas de estas presentaban hifas y zoosporangios de oomicetos del género Lagenidium tanto en el tejido exterior como en los tejidos interiores (Fig. 2). El hecho de que Lagedinium sp. no fue observado en todas las zoeas muertas nos indica que este no fue el factor principal en la muerte de estas.
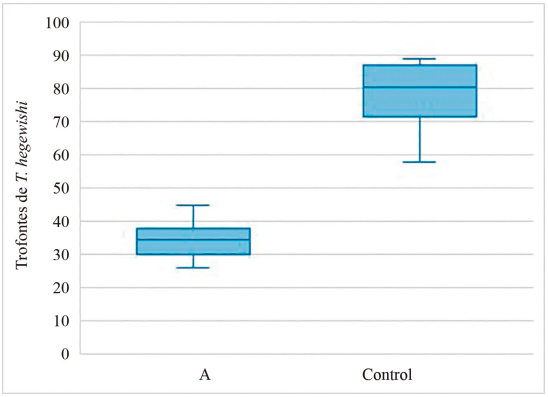
Los resultados de la prueba t de Student (Fig. 3) muestran una diferencia significativa entre los tratamientos para la cantidad de trofontes de T. hegewishi (t = -11.53, P <0.05), teniendo el tratamiento A mayor número de individuos en relación al tratamiento control.
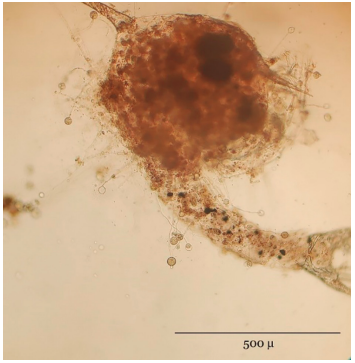
Figura 1. Trofonte de T. hegewishi alrededor de una zoea en el tratamiento A.
Figura 2. Zoea muerta de Cardisoma guanhumi del tratamiento control, con hifas y zoosporangios de Lagenidium.
Figura 3. Gráfico de cajas mostrando la cantidad de trofontes de T. hegewishi en tratamientos.
DISCUSIÓN
Según los resultados de la prueba t de Student, T. hegewishi se ve afectado por cambios repentinos en la salinidad, correspondiendo a lo expuesto por (Hwang et al., 2019), donde se asegura que asegura que la cantidad de células en los cultivos de Tetrahymena aumentan en bajas concentraciones de salinidad. En algunos estudios, la mortalidad de Tetrahymena se atribuye a la ineficacia de las vacuolas contráctiles para controlar la presión osmótica tras el aumento de la salinidad (Ayre et al., 2017); sin embargo, otros niegan el efecto de la salinidad en la formación de estos orgánulos (Carnegie et al., 2019).
La presencia de oomicetos es una variable a considerar en la mortalidad de las larvas ya que, aunque no se haya observado en todas las zoeas, algunos autores informan que algunas especies están exclusivamente relacionadas con cangrejos, tendiendo a colonizar la superficie del individuo desde donde dispersa sus zoosporas (Lee et al., 2016). En algunos casos, se han observado las hifas de Lagenidiun sp. propagarse rápidamente en los cultivos de cangrejos afectando hasta el 100 % de los huevos y las zoeas ( Van West y Beakes, 2014; Selvi et al., 2019).
Cabe destacar que anteriormente han sido mencionadas relaciones interespecíficas entre ciliados y oomicetos. Se ha reportado competencia entre ambos organismos, siendo el oomiceto causante de bajas en la densidad del ciliado (Canter y Dick, 1994). También se ha descrito mutualismo, ya que en algunas ocasiones los ciliados sirven como agentes dispersores de propágulos del oomiceto (Larousse y Galiana, 2017). A diferencia de T. hegewishi, algunos representantes del género Lagenidiun tienden a presentar crecimiento activo en salinidades con un rango tope de 30 ppm, por lo que no sorprende su presencia en cantidades similares en ambos tratamientos (Lee et al., 2016).
CONCLUSIÓN
Una solución temporal, para reducir los eventos depredadores de T. hegewishi a larvas de C. guanhumi en los cultivos, es realizar cambios repentinos en la salinidad. Un punto a considerar es el hecho de que C. guanhumi es un organismo eurihalino, que soporta cambios repentinos en la salinidad (Giménez-Hurtado et al., 2015). Un aumento gradual de la salinidad hasta la erradicación de los trofontes de T. hegewishi podría ser una alternativa. Sin embargo, una posible proliferación de oomicetos puede ser un problema, ya que estos son resistentes a salinidades relativamente altas. Estas razones hacen que no se pueda asegurar el éxito del uso de cambios bruscos en la salinidad como método de desinfección. El uso de otras variables fisicoquímicas del agua puede ser una opción, pero se necesitan realizar más ensayos para determinar su eficacia.
AGRADECIMIENTOS
A Nabab Feliz y Antonia Marte por su ayuda de laboratorio. A Omar Paíno y Frank Reyes por su apoyo en la identificación del oomiceto. Asimismo, a la Comisión General del Acuario Nacional de Santo Domingo por dar la aprobación del proyecto. Un agradecimiento especial a Phamela Acosta Pérez por brindar sugerencias invaluables al manuscrito.
Literatura Citada
Ayre, J., F. Ng y D. Suh. 2017. Don’t be salty! The effects of salinity on vacuole formation of Tetrahymena thermophila. The Expedition, 7: 1-8.
Barrios Saucedo, L. M. 2008. Aspectos biológicos y ecológicos del cangrejo azul (Cardisoma guanhumi, Latreille, 1825) en la Bahía de Cispatá Caribe colombiano (Grado). Universidad de Bogotá Jorge Tadeo Lozano, 138 pp.
Bio-Rad Laboratories Inc. 2021. iCycler iQ Real Time PCR Detection System Instruction Manual. Bio-Rad Laboratories Inc., pp 146.
Canter, H. M. y M. W. Dick. 1994. Eurychasmopsis multisecunda gen. et sp. nov., a parasite of the suctorian ciliate Podophrya. Mycological Research, 98: 105-117.
Canvax Biotech SL. 2012. pSpark® TA DNA Cloning System Product Manual. Canvax Biotech SL, pp 28.
Carnegie, T., K. Donohoe, M. Puente y S. Saini. 2019. Effect of salinity on food vacuole formation in T. thermophila. The Expedition, 9: 1-23.
Chakraborty, C., P. Doss, C. Patra y S. Bandyopadhyay. 2014. DNA barcoding to map the microbial communities: current advances and future directions. Applied Microbiology Biotechnology, 98: 3425-3436.
Chang, Y., G. Liu, L. Guo, H. Liu, D. Yuan, J. Xiong, Y. Ning, C. Fu, y W. Miao. 2014. Cd-Metallothioneins in Three Additional Tetrahymena Species: Intragenic Repeat Patterns and Induction by Metal Ions. Journal of Eukaryota Microbiology, 61: 333-342.
Coyne, R. S. y M. C. Yao. 1996. Evolutionary conservation of sequences directing chromosome breakage and rDNA palindrome formation in tetrahymenine ciliates. Genetics, 144: 1479-1487.
De Oliveira-Neto, J. F., M. R. Pie, M. A. Chammas, A. Ostrensky y W. A. Boeger. 2008. Phylogeography of the blue land crab, Cardisoma guanhumi (Decapoda: Gecarcinidae) along the Brazilian coast. Journal of Marine Biology Association, 88: 1417-1423.
Giménez-Hurtado, E., Y. Garcés, Y. González y A. Hurtado. 2015. Densidad poblacional de Cardisoma guanhumi (Latreille, 1825) Crustacea: Brachyura: Gercarcinidae) en el Parque Nacional Ciénaga de Zapata, Cuba. Boletín del Centro de Investigaciones Biológicas, 49: 110-124.
Hwang, J. E., J. Kim, S. Subedi y J. Tamminga. 2019. The Effect of Salinity on the Growth Rate of Tetrahymena thermophila. The Expedition, 9: 1-15.
International Business Machines Corporation (IBM). 2017. IBM SPSS statistics - versión 25. [Programa Informático]. Portsmouth, IBM.
Jung, S. J., E. Y. Im, M. C. Strüder-Kypke, S.-I. Kitamura y P. Woo. 2011. Small subunit ribosomal RNA and mitochondrial cytochrome c oxidase subunit 1 gene sequences of 21 strains of the parasitic scuticociliate Miamiensis avidus (Ciliophora, Scuticociliatia). Parasitology Research, 108: 1153-1161.
Kaczanowski, A., C. F. Brunk y S. L. Kazubski. 2016. Cohesion of Clonal Life History, Senescence and Rejuvenation Induced by Autogamy of the Histophagous Ciliate Tetrahymena rostrata. Protist, 167: 490-510.
Larousse, M. y E. Galiana. 2017. Microbial Partnerships of Pathogenic Oomycetes. PLoS Pathogens, 13: 1-7.
Lee, Y. N., Q. Chuah y K. Hatai. 2016. Biological characteristics of an obligate marine strain Lagenidium thermophilum isolated from mud crab (Scylla tranquebarica) eggs in Sabah, Malaysia. Bulletin of the European Association of Fish Pathologists, 36: 126-131.
Lynn, D. H. y F. P. Doerder. 2012. The life and times of Tetrahymena. Methods in cell biology, 109: 9-27.
Méndez-Vilas, A. 2014. Industrial, medical and environmental applications of microorganisms:
current status and trends. Wageningen academic publisher, Wageningen, pp 693.
Nielsen, H., E. M. Simon y J. Engberg. 1985. Updating rDNA Restriction Enzyme Maps of Tetrahymena Reveals Four New Intron-Containing Species. The Journal of Protozoology, 32: 480-485.
Nyberg, D. 1981. Three New “Biological” Species of Tetrahymena (T. hegewischi n. sp., T. sonneborni n. sp., T. nipissingi n. sp.) and Temperature Tolerance of Members of the “pyriformis ” Complex1. The Journal of Protozoology, 28: 65-69.
Nzytech. 2021. NZYProof DNA polymerase | DNA Polymerases, End-Point PCR. NZYProof DNA polymerase.
Qiagen. 2020a. DNeasy Blood & Tissue Kits Handbook. Qiagen, pp 64.
Qiagen. 2020b. QIAprep® Miniprep Handbook. Qiagen, pp 57.
Roche Sequencing. 2021. KAPA dNTPs. KAPA dNTPs. (/content/rochesequence/en/productssolutions/products/pcr/kapa-dntps.html).
Ruehle, M. D., E. Orias y C. G. Pearson. 2016. Tetrahymena as a Unicellular Model Eukaryote: Genetic and Genomic Tools. Genetics, 203: 649-665.
Schlegel, M. 1991. Protist evolution and phylogeny as discerned from small subunit ribosomal RNA sequence comparisons. European Journal of Protistology, 27: 207-219.
Selvi, M., S. Afandi y U. A. Maria. 2019. Effect of Antifungal Treflan And Lentil Essential Oil Disc Inhibition Methods On Lagenidium Callinectes. Russian Journal of Agricultural and Socio-Economic Sciences, 89: 27-31.
Sigma-Aldrich. 2021. Sanger Sequencing Steps | DNA Sequencing. Sigma-Aldrich. (https://www.sigmaaldrich.com/technical-documents/articles/biology/sanger-sequencing.html).
Thermo Fisher Scientific Inc. 2010. NanoDrop ND-1000 Spectrophotometer V3,8 User’s MAnual.
Untergasser, A., H. Nijveen, X. Rao, T. Bisseling, R. Geurts y J. A. M. Leunissen. 2007. Primer3Plus, an enhanced web interface to Primer3. Nucleic Acids Research, 35: 71-74.
U.S. National Library of Medice. 2021. Nucleotide BLAST: Search nucleotide databases using a nucleotide query. Blast. (https://blast.ncbi.nlm.nih.gov/Blast.cgi?PAGE_TYPE=BlastSearch).
Van de Peer, Y., J.-M. Neefs, P. De Rijk y R. De Wachter. 1993. Evolution of eukaryotes as deduced from small ribosomal subunit RNA sequences. Biochemical Systematics and Ecology, 21: 43-55.
Van West, P. y G. W. Beakes. 2014. Animal pathogenic Oomycetes. Fungal Biology, 118: 525-526.
Washburn, J. O., D. E. Egerter, J. R. Anderson y G. A. Saunders. 1988. Density reduction in larval mosquito (Diptera: Culicidae) populations by interactions between a parasitic ciliate (Ciliophora: Tetrahymenidae) and an opportunistic fungal (Oömycetes: Pythiaceae) parasite. Journal of medical entomology, 25: 307-314.
Wilkes, D. E., N. Bennardo, C. W. C. Chan, Y.-L. Chang, E. O. Corpuz, J. DuMond, J. A. Eboreime,
J. Erickson, J. Hetzel, E. E. Heyer, M. J. Hubenschmidt, E. Kniazeva, H. Kuhn, M. Lum, A. Sand, A. Schep, O. Sergeeva, N. Supab, C. R. Townsend, L. V. Ryswyk, H. E. Watson, A. E. Wiedeman, V. Rajagopalan y D. J. Asai. 2009. Identification and Characterization of Dynein Genes in Tetrahymena, pp. 11-30. In Methods in Cell Biology. Elsevier.
Xiong, J., W. Yang, K. Chen, C. Jiang, Y. Ma, X. Chai, G. Yan, G. Wang, D. Yuan, Y. Liu, S. L. Bidwell, N. Zafar, M. Hadjithomas, V. Krishnakumar, R. S. Coyne, E. Orias y W. Miao. 2019. Hidden genomic evolution in a morphospecies-The landscape of rapidly evolving genes in Tetrahymena. PLoS Biology, 17: 2297-2302.
Ye, A. J. y D. P. Romero. 2002. Phylogenetic relationships amongst tetrahymenine ciliates inferred by a comparison of telomerase RNAs. International Journal of Systematic and Evolutionary Microbiology, 52: 2297-2302.